Naukowcy z UMD i NIST odkryli sposób wykrywania mechanizmu molekularnego, za pomocą którego aktywowany jest 5HT3A, receptor serotoninowy zlokalizowany w synapsie neuronu.
Posiadanie modelu molekularnego tej aktywacji pozwala na testowanie inhibitorów farmaceutyków przy użyciu modeli komputerowych zamiast tradycyjnych eksperymentów. To z kolei potencjalnie prowadzi do zmniejszenia kosztów i potrzebnego czasu na badania nowych leków.
Badanie było prowadzone przez Jeffery Klauda, profesora inżynierii chemicznej i biomolekularnej (ChBE) na University of Maryland (UMD). Z kolei Nicholas Guros, asystent naukowy ChBE, był pierwszym autorem badań, któremu w pracy pomógł Arvind Balijepalli, inżynier mechanik w NIST. Samo badanie zostało z kolei opublikowane w Proceedings of National Academy of Sciences (PNAS).
5HT3A jest rodzajem białka zwanego kanałem jonowym, powiązanym z depresją, lękiem i ukierunkowanym na zmniejszenie nudności wywołanych chemioterapią. Reguluje przepływ jonów w post-synaptycznym rozszczepie neuronów.
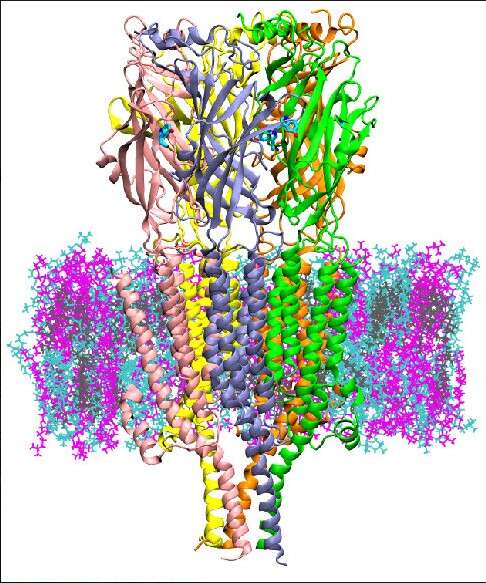
Autorzy badania przeprowadzili symulacje komputerowe 5HT3A przez maksymalnie 20 mikrosekund. To znacznie dłużej w porównaniu do jakichkolwiek poprzednich symulacji. Aktywacja i odczulanie białek, takich jak 5HT3A, zachodzi w ciągu milisekund, więc naukowcy wciąż nieco opóźniają obserwację całego cyklu transportu białka.
Badanie to było jednak w stanie zapewnić wgląd w wpływ wiązania serotoniny na rozwój stanu preaktywnego 5HT3A oraz tego, w jaki sposób różnorodność lipidów błonowych wpływa na strukturę i funkcję białka.
Wymaga to jednak udziału superkomputera i dlatego wraz ze wzrostem prędkości i mocy obliczeniowej w przyszłości grupa ma nadzieję zbadać działanie farmaceutyków, takich jak granisetron lub ondansetron, w celu dokładnego modelowania ich wpływu na białko i zbadania innych ukierunkowanych terapii.