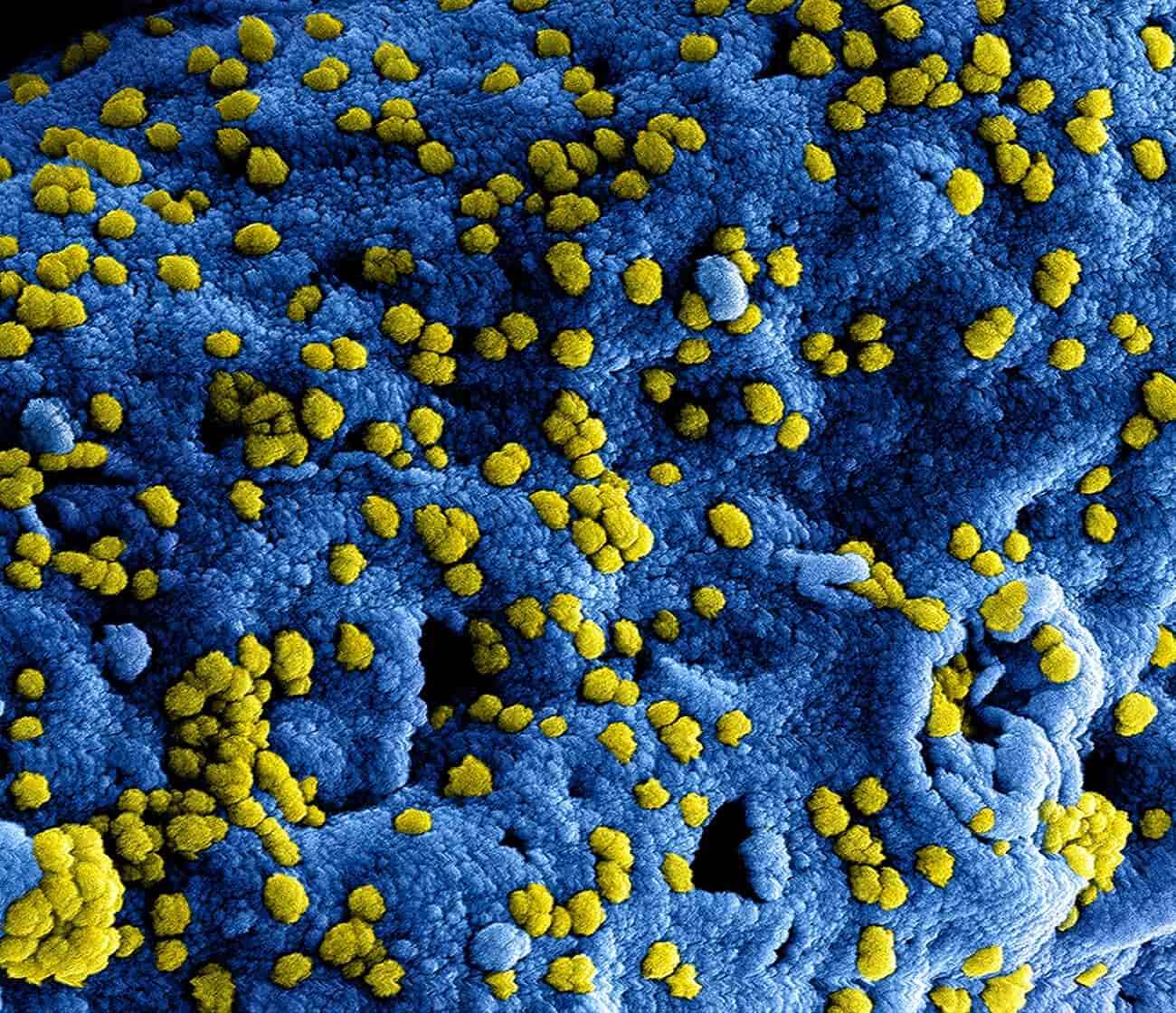
Niemal dwadzieścia lat temu po raz pierwszy koronawirus ujawnił swój pandemiczny potencjał, choć ani SARS ani MERS nie rozprzestrzeniły się po świecie.
Niestety, w przypadku SARS-CoV-2, czyli patogenu wywołującego COVID-19, nie mieliśmy tyle szczęścia. Wykorzystując uczenie maszynowe naukowcy postanowili określić, które ssaki mogą być nosicielami wielu szczepów koronawirusa, aby oszacować, czy (a w zasadzie kiedy) może wystąpić kolejna pandemia.
Czytaj też: Bill Gates sądzi, że walka z pandemią jest łatwa w porównaniu z tym, co nas czeka
Czytaj też: Czy przeziębienie zapewnia ochronę przed COVID-19?
Czytaj też: Pandemia doprowadziła do wyludnienia miast, ale co dalej?
Artykuł w tej sprawie, dostępny w Nature Communications, sugeruje, iż znaleziono ponad 30 razy więcej potencjalnych żywicieli, którzy mogliby być siedliskiem SARS-CoV-2 i doprowadzić do jego rekombinacji. Z kolei liczba gatunków, które wcześniej podejrzewano o bycie żywicielami kilku podrodzajów koronawirusa okazała się 40-krotnie wyższa.
Nie wiadomo, kiedy wystąpi kolejna pandemia, ale zagrożenie jest niedoceniane
I choć naukowcy z Uniwersytetu w Liverpoolu nie są w stanie ocenić, gdzie i kiedy rozpocznie się kolejna pandemia, to z badania płynie jasny wniosek: należy zmienić podejście do całej sprawy. Mowa zarówno o przewidywaniu i prowadzeniu badań, ale również o podjęciu zdecydowanych działań mających na celu ograniczenie ryzyka „przeskakiwania” patogenów z dzikich zwierząt na ludzi.
Czytaj też: Szczepionka AstraZeneca może zahamować pandemię COVID-19. Skąd więc kontrowersje?
Czytaj też: Kolejny sezon grypowy może być znacznie cięższy. Wszystko przez pandemię
Czytaj też: Jak limfocyty T chronią nas przed COVID-19?
Dotychczasowe obserwacje pozwalały sądzić, że istnieją tylko cztery ssaki inne od ludzi, które mogą być nosicielami zarówno SARS-CoV-2, jak i co najmniej jednego innego koronawirusa. W praktyce jednak istnieje minimum 126 nosicieli SARS-CoV-2 oraz 2544 interakcji, które mogłyby doprowadzić do powrotu tego patogenu w jeszcze groźniejszej wersji.